
Multiplex-Polymerase Chain Reaction을 이용한 대마의 조기 성판별 Sequence Characterized Amplified Region 마커 개발
This is an open access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
Cannabis are typically dioecious, with female and male reproductive parts located on separate plants. Female cannabis plants are preferred for the production of cannabinoids, terpenes, and other valuable compounds synthesized within the female floral tissue. Female and male plants look identical during seedling and vegetative phases, and only after transition to flowering phase can male, female, and monoecious plants be distinguished. Early identification of the sex of cannabis plant can conserve time and money by avoiding the undesired sex of the plant. The objective of the present study was to provide a sex determination method for cannabis plants using the sequence characterized amplified region (SCAR) marker, and set multiplex-polymerase chain reaction (PCR) conditions for more accurate early sex determination of cannabis to overcome the disadvantages of SCAR markers.
Cannabis seeds were collected and cultivated for several purposes. Leaf samples were harvested and stored during vegetative growth phase. In silico mapping of three male-specific sequences (MADC3, MADC4, and MADC6) revealed through random amplified polymorphic DNA, showed that MADC3 has a very similar sequence with the female cannabis genome. Primers were produced based on the sequences (SCAR1, SCAR2, and SCAR3, respectively) and subjected to gradient PCR. Only SCAR3 produced a male-specific band with an annealing temperature of over 61.4℃. PCR was performed on cannabis plants produced for several purposes by mixing SCAR3 and internal standard 3 (IS3). Result showed that all cannabis plants produced an IS3 band of 197 bp, but only male cannabis plants produced a SCAR3 band of 118 bp.
Multiplex-PCR primer produced a male-specific band of 119 bp in cannabis bred for several purposes, and the presence of the 197 bp cannabis common band confirmed that PCR proceeded normally for all samples. The multiplex-PCR primer is greatly beneficial because the IS3 band is a specific sequence found only in cannabis, making it possible to differentiate cannabis from other plants in addition to determining the sex from with a single PCR run.
Keywords:
Cannabis sativa L., Hemp, Marijuana, Sequence Characterized Amplified Region Marker, Sex Identification, Multiplex Polymerase Chain Reaction서 언
대마 (Cannabis sativa L.)는 고대부터 중앙아시아에서 주로 재배되어지는 1년생 초본으로, 이배체 (2n = 20)의 핵형을 가지고 있고, 드물게 자웅동주 (雌雄同住)가 발견되어지지만 일반적으로 자웅이주 식물의 특성을 가지고 있다 (Russo et al., 2008).
고대 중국에서 섬유를 얻을 목적으로 재배된 기록을 기반으로 점차 재배량이 늘어난 대마는 (Zhang et al., 2020), 10,000년 이상 동안 의료의 목적으로 이용되었다 (Schultes et al., 1975)
대마는 525 종 이상의 화합물을 함유하고 있으며, 그중 약 109 종 이상의 C21 terphenophenol 계열 화합물인 칸나비노이드를 함유하는 것으로 알려져 있다 (de Backer et al., 2012). 칸나비노이드 중 cannabidiol (CBD)와 Δ9-tetrahydrocannabinol (Δ9-THC)는 가장 잘 알려져 있으며, 대마의 이용에 있어 통상적인 재배의 목적이 되는 성분이다 (Boggs et al., 2018).
CBD는 비 향정신성 칸나비노이드로, in vitro 및 in vivo에서 신경보호 (Crippa et al., 2018), 간질에서의 강력한 항경련효과 (Consroe and Wolkin, 1977), 항불안 (Guimarães et al., 1990), 항정신병 (Chesney et al., 2022), 항염증 (Atalay et al., 2019), 진통 (Urits et al., 2020) 및 항암효과 및 항암제와의 상승작용을 가지는 것으로 알려져 있다 (Go et al., 2020; Koltai and Shalev, 2022; Tajik et al., 2022).
또한, Δ9-THC은 향정신성 칸나비노이드로, 대부분의 국가에서 마약류로 관리되어지고 있으며 해당 성분은 투여 시 일시적인 불안을 유발하며 (Hindley et al., 2020), 학습능력 및 기억력 저하 (D'Souza et al., 2005), 편집증 및 우울증 (Freeman et al., 2015)을 유발하는 등의 부작용을 나타낸다.
이러한 두 종류의 주된 칸나비노이드의 95% 이상은 식물체 내에서, CBD는 CBDA (cannabidiolic acid), Δ9-THC는 Δ9-THCA (Δ9-tetrahydrocannabinolic acid)의 카르복실기가 결합되어져 있는 형태로 존재하며 (Tahir et al., 2021), 가열 처리에 의해 카르복실기가 해리되어 활성을 나타내는 형태로 전환되어진다 (Ryu et al., 2021).
칸나비노이드는 영양 생장 시기에도 식물체 내에서 미량의 수준으로 합성되기도 하지만, 자성 (雌性, female) 개체 꽃받침 부분의 모용 (trichome)에서 높은 수준으로 생산되어지기때문에 (Rodziewicz et al., 2019), 모용이 존재하지 않는 웅성 (雄性, male) 개체에서의 칸나비노이드 생산은 자성 개체에 비하여 극히 적다 (Fig. 1; Small and Beckstead, 1973; Mansouri et al., 2013). 웅성 개체의 화아 분화는 환경에 따라 달라질 수 있지만 대개 암꽃의 출현보다 대략 1 개월 정도 빠르며, 수꽃 (雄性花, male flower)에서 비산되는 화분 (pollen)이 재배환경에서 부유하다가 암꽃 (雌性花, female flower)에 수정되어지면, 자성 개체의 CBD와 Δ9-THC의 비율은 그대로 유지되나 칸나비노이드의 절대적인 전체 함량은 감소한다고 알려져 있으며 (ElSohly et al., 2017), 이러한 특성때문에 상업적으로 생산되는 대마 식물체는 자성 개체만을 대상으로 하고 있다.

Morphological shape of male and female cannabis.(A); male cannabis, (B); female cannabis. Micrograph shows the trichome of a female cannabis inflorescence. Typically, female trichome contains higher levels of cannabinoids and terpenes concentration than normal tissue.
대마를 포함한 자웅이주 식물체에서 성별 결정은 성염색체, DNA 메틸화와 miRNA에 의한 후생적 조절, 식물 호르몬에 의한 생리학적 조절 등 여러 요인에 의해 좌우되며 (Aryal and Ming, 2014; Heikrujam et al., 2015; Bai et al., 2019), 잘 정의된 이형 염색체를 가지고 있음에도 불구하고 환경적 요인의 변화에 따라 대마의 성별이 결정될 수 있다 (Schaffner, 1921).
현재까지 aminoethoxyvinyl glycine (AVG, C6H12N2O3)등의 에틸렌의 생합성이나 활성을 억제하는 화학물질 처리는 대마의 수꽃을 유도하며, ethephon (C2H6ClO3P)과 같은 에틸렌 생합성 전구체는 암꽃의 발생을 촉진시키는 효과가 있다고 알려진다 (Mohan Ram and Sett, 1982a; 1982b). 이러한 화학적 처리에 따른 성별 표현형의 전환은 품종에 따라 화학적 처리제의 종류와 농도에 따라 반응하는 감응도가 서로 다른 것으로 알려져 있다 (Moliterni et al., 2004).
자성 종자화는 외생의 호르몬 처리를 통하여, 자성 개체 (XX)에서 성별 표현형을 전환시켜 수꽃을 유도하고, 이들이 생산한 꽃가루를 자성 개체의 암꽃에 수분시켜 자성 개체로 유전형이 고정된 종자를 생산하는 방식이다. 최근의 대마 육종에서 육종가들은 칸나비노이드 함량을 극대화하기 위하여 silver nitrate (AgNO3) 및 sodium thiosulfate (Na2S2O3) 혼합처리를 통해 목적으로 하는 화학형 (chemotype) 집단의 자성 개체에서 수꽃을 유도하여 육종 및 품종 고정화를 진행하고, 높은 동형접합성의 자성 종자를 공급함으로써 카나비노이드를 고수율로 생산하는 방식을 택하고 있다 (Salentijn et al., 2015; Lubell and Brand, 2018; Flajšman et al., 2021).
하지만, 식물체 특성상 화아 분화가 일어나기 전의 자성 및 웅성 개체의 대마는 형태적으로 매우 유사하며, 자성 및 웅성 개체 모두 미량의 칸나비노이드를 함유하기 때문에 성별의 조기 식별이 어려운 실정이다 (ElSohly et al., 2017). 이는 자성 및 웅성 개체가 혼입되어있는 상태에서 서로 다른 칸나비노이드 함량에 근거하여 원료의약품 품질 표준화에 제한이 되고 있으며, 재배 중, 유전적인 혼입이 일어날 수 있는 수정을 방지하기 위해 웅성 개체를 조기 선발하여 제거하는 것이 필 요하다.
Lacombe (1980)는 형태학적 특성을 기반으로 대마의 성별 상관관계를 분석하여 특정화를 시도하였으며, 추후 분자 마커가 개발된 이후 대마의 성 판별이 더 정밀하게 가능하게 되었다. 분자 마커는 생물이 가지고 있는 DNA 염기서열을 기반으로 종 혹은 집단의 특성을 비교 분석하는 방법으로 (Kim et al., 2021), random amplified polymorphic DNA (RAPD), amplified fragment length polymorphism (AFLP), inter-simple sequence repeat (ISSR) 등이 사용되어왔으며, 최근에는 임의 증폭 마커 기반에서 기능성 유전자 타겟 마커인 conserved DNA-derived polymorphism (CDDP), sequence related amplified polymorphism (SRAP), start codon targeted polymorphism (SCoT) 등이 사용되어지고 있다 (Huh, 2015).
상추의 병저항성 유전자의 발굴을 위해 처음 개발된 sequence characterized amplified region (SCAR) 마커는 (Paran and Michelmore, 1993), 사전 염기서열이 요구되지 않는 RAPD, ISSR 및 AFLP 등의 유전 분석 기술보다 재현성과 신뢰성 및 특이성이 높아 식량작물 (Naqvi and Chattoo, 1996; Ohmori et al., 1996; Maliepaard et al., 1998; Gupta et al., 2006; Khampila et al., 2008) 및 약용작물 (Lim et al., 2007; Park et al., 2007; Jo et al., 2017; Kim et al., 2022)에서 활발히 사용되어지고 있다.
하지만 특이적인 우성 마커 특성상 PCR amplicon band의 유무에 따라 개체의 변이를 판별하기 때문에 실험 과정에 비롯된 유전자 증폭에 관한 오류인지 혹은 증폭 DNA loci의 결손에 따른 음성 판정의 결과인지에 대한 검증이 필수적으로 필요하다 (Xu et al., 2006)
따라서 본 연구에서는 대마의 웅성 특이적으로 알려져 있는 3 종의 RAPD 기반 염기서열을 In silico 조건에서 유효성을 평가하며, SCAR 마커를 디자인하여 in vitro 조건에서 의료용 대마, 마약성 대마, 섬유용 대마 및 야생형 대마를 대상으로 실제 적용하여 범용성 여부를 평가하였다. 또한 2 쌍 이상의 프라이머 조합을 사용한 multiplex-PCR을 기반으로, 성별 특이적 서열과 대마에서 공통으로 발현되어지는 internal standard (IS) 서열을 동시에 증폭시켜, 더욱 정확한 대마 영양생장기의 성 판별을 위한 SCAR 마커의 PCR 분석법을 확립하고자 하였다.
재료 및 방법
1. 식물재료 및 DNA 추출
대마 (Cannabis sativa L.)의 종자는 야생형 대마 (인도, 영국, 이탈리아, 포르투갈 재래종 및 육종 과정 중 얻어진 대마), 국내 섬유용 대마 (청삼), 의약품 제조를 위한 원료로 사용되어지는 의료용 대마 (Cinderella story, Cherry blossom, Cloud berry) 및 마리화나로 분류되는 화학형을 가진 대마 (Critical +2.0)를 사용하였으며, 한국대마유전자원센터 (Chuncheon, Kroea)에서 제공받아 사용하였다.
수집된 종자는 온도 23 ± 2℃, 상대습도 50 ± 5% 조건의 배양실에서 상토 (AsiaSeedKorea Co., Seoul, Korea)에 2 ㎝ 깊이로 파종하였으며, 2 일 간격으로 관수하여 재배하였다. 영양생장기 중 식물체의 잎을 상, 중, 하단부에서 미리 채취한 뒤, -81℃의 deep freezer (DAIHAN Scientific Co., Ltd., Wonju, Korea)에서 보관하였다.
파종 후 2 달간 식물의 영양 생장을 유도하기 위하여, 적색 및 청색으로 이루어진 light emitting diode (LED, BISSOLLED, Seoul, Korea)를 사용하여 약 312.9 ± 0.5 µmol/m2·s의 photosynthetic photon flux density (PPFD) 조건으로 광주기를 18 h/day으로 조정하여 광을 조사하였다.
파종일 기준 2 개월 동안 영양 생장이 이루어졌으며 생식생장을 유도하기 위하여 광주기를 12 h/day의 단일처리를 통하여 화아분화를 유도하였다. 개화 시기의 PPFD 값의 증가는 화서의 정량적 수율을 증가시킨다는 보고를 (Rodriguez-Morrison et al., 2021) 토대로 하여, 화서의 분화를 촉진시키고, 대마의 성별 식별을 용이하게 하기 위하여 생식 생장을 위한 단일 처리 시 PPFD 값을 약 597.4 ± 1.9 µmol/m2·s 수준으로 2 배 증가시켰다 (Table 1). 작물이 생육함에 따라 초장의 변화를 나타내기 때문에, 자체적인 광원 거리와 PPFD 간의 calibration curve를 생성하였고, 도출된 회귀방정식 (식 1)에 대입하여 전체 생육기간동안 동일한 수준의 광량이 조사 될 수 있도록 하였다.
| (식 1) |
화아분화가 유도되어 암꽃 혹은 수꽃이 출현하였을 때, 꽃을 경시적으로 관찰하여 자성개체와 웅성개체를 구분하였으며 미리 채집된 대마 잎을 대상으로 하여 DNA를 추출하였다.
영양생장 시기에 미리 채집된 대마의 각 잎은 -196℃의 액체질소 및 막자사발을 사용하여 분쇄 및 균질화시켰으며, DNA extraction kit (i-genomic Plant DNA Extraction Mini Kit, iNtRON Biotechnology Inc., Sungsam, Korea)을 이용하여 제조사의 프로토콜을 따라 DNA를 추출하였고 추출된 gDNA는 미량 분광분석기 (Optigen NanoQ, KLAB, Daejeon, Korea)를 이용하여 70 ng/㎕로 희석시켜 사용하였다.
2. 프라이머
대마 조기 성 판별용 SCAR 마커의 프라이머는 RAPD를 통하여 밝혀진 대마 웅성 개체 특이적 염기서열 정보 3 종인 MADC3 (GenBank accession number: AB021658.1; Sakamoto et al., 2005), MADC4 (GenBank accession number: AB021659.1; Sakamoto et al., 2005), MADC6 (GenBank accession number: AF364955.1; Törjék et al., 2002)를 기반으로 (Fig. 2), CLC Genomics Workbench (v. 22.0.2, QIAGEN, Hilden, Germany) 프로그램을 사용하여 설계하였다.

DNA sequence for cannabis gender determination used in this study.The red-highlighted regions represent the base sequences of the forward and reverse primers used to amplify the target DNA. A; MADC3 (Sakamoto et al., 2005; GenBank: AB021658.1; 769 bp), B; MADC4 (Sakamoto et al., 2005; GenBank: AB021659.1; 576 bp), C; MADC6 (Törjék et al., 2002; GenBank: AF364955.1; 151 bp).
프라이머 설계 시 염기서열 양쪽 말단의 60 bp를 기준으로 설정하였으며, 프라이머의 길이는 18 - 22 bp, G/C 함량은 0.4 - 0.6, melting temperature (Tm)은 48℃ - 58℃으로 설정하였으며, 프라이머셋의 최대 Tm 차이는 5℃로 설정하였다. 생성된 각 서열의 프라이머 리스트 중 Multiplex Primer Analyzer (Thermo-Fisher Scientific Inc., Waltham, MA, USA)를 사용하여 IS 프라이머와 cross-dimer형성을 최대한 우회하여 디자인하였다 (Table 2).
Multiplex-PCR용 IS 프라이머는 대마 공통 tetrahydrocannabinolic acid synthase를 코딩하고 있는 CHF3/ISR 및 CHF1/ISR2 프라이머와 (Doh et al., 2019), 엽록체 trnL과 trnF 유전자 사이의 대마 유전체의 특이적인 구간을 증폭시키는 trnL-F 프라이머를 사용하였다 (Linacre and Thorpe, 1998; Table 2).
본 연구에서는 편의상 CHF3/ISR 프라이머셋은 IS1, CHF1/ISR2 프라이머셋은 IS2, trnL-F 프라이머셋은 IS3로 설정하여 실험을 진행하였다 (Table 2).
3. PCR (Polymerase Chain Reaction)
Gradient PCR은 전체 50.0 ㎕로 10 × reaction buffer 5.0 ㎕, 10 mM stabilized dNTP 1.0 ㎕, 20 µM forward 및 20 µM reverse primer 각 1.0 ㎕, 70 ng/㎕ gDNA template 2.0 ㎕, 멸균 초순수 39.5 ㎕ 및 Taq DNA polymerase 0.5 ㎕를 혼합하여 사용하였으며, Thermal Cycler Dice Touch (TP350, TAKARA bio Inc., Kusatsu, Japan)를 사용하여 94℃에서 3 분간 초기 변성과정을 거쳤으며, 94℃에서 30 초간 denaturation, 50℃ - 70℃에서 30 초간 gradient annealing, 72℃에서 60 초간 extension을 30 cycle 반복 진행하였다. 마지막으로 72℃에서 7 분간 추가 신장을 통하여 반응을 마무리 지었으며, 실험에 즉시 사용되지 않는 product는 4℃에서 보관하였다.
Multiplex-PCR은 annealing temperature를 제외하고 상기조건과 동일하였으며, 사용된 프라이머의 비율은 성판별 SCAR 마커와 Internal standard 프라이머셋을 3 : 1 비율로 추가하여 진행하였다.
4. 전기영동
PCR product의 전기영동은 molecular biology grade의 2% agarose (MP Biomedicals, Irvine, CA, USA) gel을 이용하였으며, amplicon fragment의 형광염색을 위하여 EtBr (Ethidium bromide, Sigma-Aldirch Co., St. Louis, MO, USA)을 20 ㎍/ℓ농도로 첨가하였다. TAE buffer (Lonza, Basel, Switzerland)를 채운 chamber에 agarose gel을 위치시킨 후 6× DNA loading dye (TAKARA bio Inc., Kusatsu, Japan)와 PCR product를 혼합하여 로딩한 뒤 전기영동장치 (TAKARA bio Inc., Kusatsu, Japan)를 사용하여 전기영동을 진행한 후, gel documentary system (Bio-rad Inc., Hercules, CA, USA)을 사용하여 가시화하였다. DNA ladder는 BIONEER사 (100 bp, Daejeon, Korea)의 제품을 사용하였다.
결과 및 고찰
1. SCAR 마커 평가 및 최적 온도 설정
CLC Genomics Workbench 프로그램을 사용하여 In silico에서 SCAR 마커의 기반이 되는 MADC3, MADC4 및 MADC6 서열을 자체적인 자성 의료용 대마 (2n = 20)의 chromosome 수준의 whole genome (데이터 미공개)과 mapping하여 자성 대마에서 유사 서열을 탐색한 결과, 세 가지 서열 중 MADC3 유사 서열이 의료용 자성 대마의 5 번 염색체에서 검출되었고, 해당 염색체의 15,822,646 bp - 15,823,401 bp에 존재하는 것을 확인하였다 (Fig. 3A). 의료용 대마의 genome data 상에서 해당 locus는 non-CDS (non-coding sequence)로 별도의 아미노산을 코딩하고 있지 않은 intron 부분임을 확인하였다.
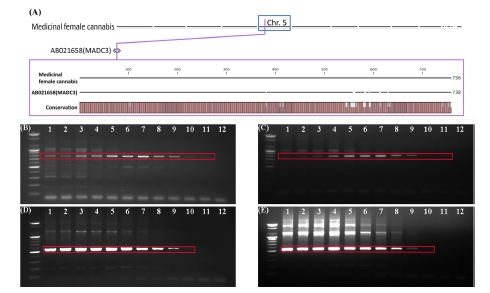
Detection of MADC3 seq. In silico and MADC3 seq., MADC4 seq. in vitro using SCAR marker.(A); whole genome alignment (mapping) results of medicinal female cannabis and MADC3 sequences, (B); gradient PCR results of ‘female’ cannabis using SCAR1 primer based on MADC3 sequence, (C); gradient PCR results of ‘male’ cannabis using SCAR1 primer based on MADC3 sequence, (D); gradient PCR results of ‘female’ cannabis using SCAR2 primer based on MADC4 sequence, (E); gradient PCR results of ‘male’ cannabis using SCAR2 primer based on MADC4 sequence. Numbers indicate PCR annealing temperature; lane 1; 50.0℃, lane 2; 50.6℃, lane 3; 51.8℃, lane 4; 53.8℃, lane 5; 56.6℃, lane 6; 58.8℃, lane 7; 61.4℃, lane 8; 63.6℃, lane 9; 66.6℃, lane 10; 68.6℃, lane 11; 69.8℃, and lane 12; 70.0℃.
MADC3 서열과 자체적 자성 대마의 whole genome은 다량의 SNP (single nucleotide polymorphism) 혹은 InDel (insertion and deletion) 변이를 포함하고 있었지만, 염기 일치구간을 총 서열 구간으로 나누어 판독한 두 서열의 유사도는 약 94%였다. 웅성 대마 특이적인 서열이라 알려진 해당 서열은 암컷 대마에서 매우 유사한 서열로 mapping 되는바, 해당 MADC3 서열은 분자 마커의 PCR 조건을 조절하여 성별을 구분할 수 있을 가능성이 매우 낮았다
또한, MADC3 서열을 기반으로 제작된 SCAR1 마커를 사용하여 gradient PCR을 진행한 결과 또한 암·수의 차이 없이 annealing temperature가 69.8℃에 이르기까지 공통으로 band 를 확인할 수 있었으며 (Fig. 3B and 3C), 해당 band는 700 bp - 800 bp로 확인되어 SCAR1 마커의 목적 amplicon size인 754 bp에 부합함을 확인할 수 있었고, 결과적으로 MADC3 서열을 기반으로 제작한 SCAR1 마커는 annealing temperature를 조절하여 대마의 성별 구분이 불가능함을 확인하였다 (Fig. 3B and 3C).
또한, In silico 환경의 암컷 대마에서 확인되지 않은 MADC4 서열을 SCAR 마커로 디자인 (SCAR2)하여 gradient PCR 조건에서 분석한 결과 암·수의 차이 없이 68.6℃의 annealing temperature에서 동일한 위치에서 band가 확인되었고, 해당 밴드는 500 bp - 600 bp로 확인되어 SCAR2 amplicon 사이즈인 555 bp에 부합됨을 확인할 수 있었고, 결과적으로 MADC4를 기반으로 만들어진 SCAR2 마커 또한 annealing temperature를 조절하여 증폭한 경우에도 암·수 구분이 불가능함을 확인하였다 (Fig 3D and 3E).
MADC3 및 MADC4 서열은 Sakamoto 등 (2005)에 의해 처음 보고된 대마 웅성 개체의 특이적 서열로, 120 여종의 random primer를 사용하여 얻어졌다. 771 bp의 MADC3 서열은 Arabidopsis thaliana, Oryza sativa, Zea mays 및 Glycine max와 같은 다양한 식물에서 copialike retrotransposon의 gag/pol protein을 암호화하는 프레임과 상동적인 것으로 분석되었으며, 540 bp의 MADC4는 아미노산 수준에서 A. thaliana 및 O. sativa의 class 1 transposable elemnts 서열과 유사하다 보고되었다.
Sakamoto 등 (2005)의 선행연구에서 두 서열은 RAPD를 기반으로 대마의 웅성 개체의 특이적 서열로서 관찰되었지만 본 연구에서는 그 재현성이 낮음을 확인할 수 있었다. 이는 선행연구에서 본 서열들이 cannabidiol을 고수율로 생산하기 위한 품종 육성에 있어 대상 계통이 제한집단으로 구성되었기 때문에, 해당 집단 내 genetic pool로 한정되어 있고 다양성이 확보되지 않은 결과로 생각되어진다. Toth 등 (2020) 또한 MADC3 및 MADC4 서열 기반의 RAPD 분자 마커는 구분하기 위한 각 개체의 암·수 구분에 대한 재현성을 확인하기 어렵다고 언급한바, 결과적으로 본 연구에서 사용한 다양한 화학형과 유전형을 가지고 있는 개체의 경우 암·수 구분을 위해 MADC3 및 MADC4 서열을 이용하는 것은 부적절할 수 있다고 판단되었다
MADC6 서열은 In silico에서 자성 개체 대마와의 유사 서열을 찾을 수 없었으며, 해당 서열을 기반으로 한 SCAR3 마커를 사용한 gradient PCR 결과, 자성 개체 대마의 목적 band는 61.4℃까지의 annealing temperature에서 검출되었으며, 수컷 대마의 목적 밴드는 69.8℃까지의 annealing temperature에서 검출되었고 (Fig. 4A and 4B), 해당 band는 118 bp인 SCAR3 마커의 목적서열에 해당함을 알 수 있었다.
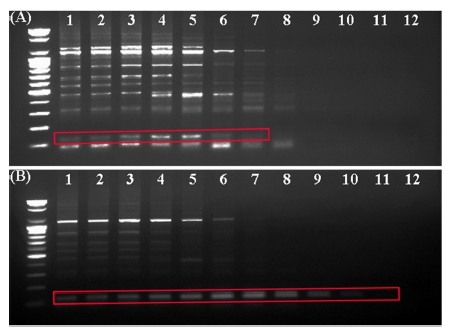
Gradient PCR results using the SCAR3 primer set based on MADC6 sequence.A, PCR result of ‘female’ cannabis; B, PCR result of ‘male’ cannabis. Numbers indicate PCR annealing temperature; lane 1; 50.0℃, lane 2; 50.6℃, lane 3; 51.8℃, lane 4; 53.8℃, lane 5; 56.6℃, lane 6; 58.8℃, lane 7; 61.4℃, lane 8; 63.6℃, lane 9; 66.6℃, lane 10; 68.6℃, lane 11; 69.8℃, and lane 12; 70.0℃.
따라서 웅성 개체의 특이적 band가 검출되어지는 annealing temperature는 61.4℃보다 높아야 하고, 69.8℃ 이하로 조정하여야 함을 알 수 있었고, 해당 MADC6 서열을 기반으로 제작된 SCAR3 마커를 이용하는 경우 annealing temperature 조건 변화에 의하여 대마 개체의 암·수 구분, 성별에 대한 조기판별 마커로 사용할 수 있음을 확인하였다.
또한, 자성 개체 대마에서는 annealing temperature 63.6℃까지 100 bp 이하의 self-dimer band가 검출되어 혼동을 야기할 수 있어 이를 최소화하고, 목적 band의 검출을 명확히하기 위하여 세부적인 온도 조건 조절을 통해 최종 annealing temperature를 65.0℃로 설정하였다.
해당 서열을 기반으로 한 분자마커는 Salix viminalis L.의 성별 구분을 위해 제작된 RAPD 마커로 처음 알려졌으며 (Alstrom-Rapaport et al., 1998), 이를 대마에 적용하여 얻어진 서열이 MADC6이다 (Törjék et al., 2002).
S. viminalis에서 해당 마커를 PCR한 결과, 자성 개체에서만 특이적으로 560 bp의 amplicon을 생산하였으며, 해당 amplicon의 sequence는 알려져 있지 않다 (Alstrom-Rapaport et al., 1998). Törjék 등 (2002)은 해당 RAPD 마커를 대마에 적용하였을 때, 마커가 기원한 식물체인 S. viminalis와 상반되게 151 bp의 비교적 짧은 서열로 이루어진 웅성 개체 특이적 amplicon인 MADC6 서열을 생산하는 것을 확인하였고, 본 연구에서 해당 서열을 기반으로 만들어진 SCAR3 마커 또한 대마의 암·수 구분에 있어 PCR 조건을 조절하여 증폭함으로서 달성될 수 있을 것으로 판단되어, 해당 SCAR3 마커를 최적의 조기 성판별 SCAR 마커에서의 프라이머로 채택하였다.
2. Multiplex PCR을 위한 Internal standard 프라이머셋의 평가 및 증폭 조건 확립
SCAR3 마커의 증폭을 위해 설정된 PCR 조건을 사용하여 Table 3의 Internal standard (IS) 마커 3 종을 야생형 대마 (인도, 영국, 이탈리아, 포르투갈 재래종 및 육종 과정 중 얻어진 대마), 국내 섬유용 대마 (청삼), 의약품 제조를 위한 원료로 사용되어지는 의료용 대마 (Cinderella story, Cherry blossom, Cloud berry) 및 마리화나로 분류되는 화학형을 가진 대마 (Critical +2.0) 품종을 사용하여 단일 증폭시킨 결과, SCAR3 마커의 PCR 조건을 사용하여 증폭시켰을 때, IS1 set은 밴드가 모든 시료에서 검출되지 않았으며, IS2 set은 각 대마 시료에서 100 bp 이하의 dimer band만 검출되었다.
하지만 IS3 set의 경우 SCAR3 프라이머를 위해 확립된 조건 하에서 암·수 공통으로 약 200 bp의 단일 밴드가 검출되었으며, 이는 197 bp의 목적 amplicon으로 판단되었다 (Fig. 5). 또한, IS1과 IS2 프라이머는 선별된 SCAR3 마커와 목적 amplicon size가 유사하기 때문에 (각 100 bp, 121 bp) SCAR3 마커와 동시 증폭이 가능하더라도 구별이 어려울 것이라 예상된다. 따라서 multiplex-PCR용 최적의 internal standard 프라이머셋을 IS3 프라이머셋으로 설정하여 추가적 실험을 진행하였다.
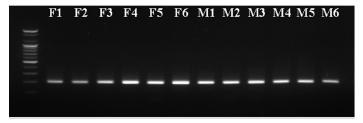
Band of the internal standard 3 (IS3) marker using the PCR condition of the SCAR3 marker.F; female cannabis, M; male cannabis. F1; Δ9-THC high variety, F2; CBD high variety, F3, CBD high variety, F4; CBD high variety, F5; fiber type variety, F6; India traditional cannabis, M1; fiber type variety, M2; England traditional cannabis, M3; Portugal traditional cannabis, M4; cannabis was randomly selected during breeding; M5; Italy traditional cannabis, M6; India traditional cannabis.
프라이머 제작 시 3 종류의 IS 프라이머셋은 유사한 이론적 annealing temperature값을 가지고 있지만, IS3 프라이머셋은 SCAR3 마커가 적용되어져야 하는 높은 annealing temperature에서도 해당 부위를 특이적으로 증폭하여 band를 형성 할 수 있었다. 이는 해당 IS3 프라이머셋이 엽록체 DNA (cpDNA)의 large unit 중앙부에 위치하는 trnL-trnF 영역을 목적으로 하며, 해당 영역은 유전자의 배열, 크기 및 구조가 동일 종 내에서 매우 보존적이며, 변이가 거의 없다는 사실을 기반으로, 제작된 프라이머가 높은 annealing temperature에서도 특이적으로 목적 locus에 접합되었을 가능성을 나타낸다 (Ryu et al., 2017).
또한, 선행연구에서 IS1과 IS2 프라이머셋의 최적 PCR annealing temperature는 54℃로 보고된 점과 (Doh et al., 2019), IS3 프라이머셋의 최적 PCR annealing temperature가 60℃로 보고된 점 (Linacre and Thorpe, 1998)을 고려할 때, multiplex-PCR에서 MADC6 서열을 증폭하기 위해 적용되는 65℃의 높은 annealing temperature의 조건에서도 IS3 프라이머셋에 의한 증폭이 가능하여 특이적인 band를 확인할 수 있었다고 판단되어진다
3. Multiplex PCR을 활용한 대마 조기 성 판별
확립된 SCAR3 마커 및 IS3 프라이머셋을 각각 3 : 1 비율로 혼합하여, PCR을 진행한 결과는 그림 6과 같다. 자성개체 대마로 확인된 F (female) 라인에서, 자성 고정형 마리화나인 Critical +2.0 (F1), 자성 고정형 의료용 대마인 Cinderella story (F2), Cherry Blossom (F3), Cloud berry (F4), 국내 섬유용 대마 청삼 (F5), 인도 자생 대마 (F6) 에서는 IS3 amplicon인 약 197 bp에서의 단일 band만 확인되었다.
반면에 웅성 개체 대마로 확인된 M (male) 라인에서, 국내섬유용 대마 청삼 (M1), 영국 자생 대마 (M2), 포르투갈 자생 대마 (M3), 육종 중 방임수정되어 무작위 선별된 웅성 개체 (M4), 이탈리아 자생 대마 (M5), 인도 자생 대마 (M6)에 서는 IS3 amplicon인 약 197 bp에서 band가 확인되었으며 조기 성 판별 SCAR3 마커의 목적 증폭서열 부분인 약 118 bp의 amplicon이 추가적으로 웅성 개체에서 확인되었다. 결과적으로, 자성 개체 대마와 웅성 개체 대마 모두 IS3 amplicon을 증폭시켜, PCR 반응이 정상적으로 진행됨을 확인할 수 있었으며, 동시에 모든 웅성 개체 대마에서 특이적으로 SCAR3 마커 amplicon이 증폭되어 암·수를 정확하게 판별할 수 있었다 (Fig. 6).
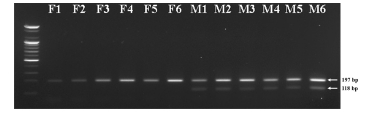
Early sex determination of cannabis using established multiplex-PCR primer sets.F; female cannabis, M; male cannabis. F1; Δ9-THC high variety, F2; CBD high variety, F3, CBD high variety, F4; CBD high variety, F5; fiber type variety, F6; India traditional cannabis, M1; fiber type variety, M2; England traditional cannabis, M3; Portugal traditional cannabis, M4; cannabis was randomly selected during breeding; M5; Italy traditional cannabis, M6; India traditional cannabis.
본 multiplex-PCR에서 사용된 IS3 프라이머셋 (trnL-F G, trnL-F H)은 높은 수준의 보존적인 cpDNA 서열 중 일부분이며, Linacre와 Thorpe (1998)의 연구에서 대마 (C. sativa)의 잎에서 추출한 gDNA만 특이적으로 197 bp amplicon이 확인되는 반면 Arabadopsis 속, Brassica 속, Zea 속 2 종, Capsicum 속, Festuca 속, Ulex 속, Humulus 속, Vicia 속, Paperva 속, Oryza 속 및 Nicotinia 속에서는 해당 위치에서의 amplicon이 확인되지 않는다고 보고된 바 있다. 또한 해당 서열과 이를 기반으로 하는 프라이머셋은 여러 속의 작물에서 유일하게 해당 위치에서 증폭됨으로서 대마 작물의 종 특이성을 확인하는 것이 가능하여 관련 서열과 프라이머 셋을 마약류 범죄에서의 DNA 기반 포렌식 기법으로 사용될 수 있다는 가능성을 제시하였다 (Linacre and Thorpe, 1998).
본 연구에서 암·수 구분을 위한 SCAR3 프라이머셋과 종특이성 평가가 가능한 IS3 프라이머셋을 이용한 multiplex-PCR 마커의 활용은 조기 성 판별의 목적인 SCAR3 마커의 정상 반응 여부를 확인함과 동시에 근연 식물이나 유사종을 조기에 식별하기 위한 용도로도 활용될 수 있을 것으로 예측 할 수 있다. 따라서 해당 multiplex-PCR 프라이머셋을 활용하여도 통상적으로 유통되어지는 식물체 중 대마만을 특이적으로 식별하는 기능을 유지하는지에 대한 여부를 평가하였다.
시중에서 판매되어지는 근대 (Beta vulgaris var. cicla), 완두 (Pisum sativum), 가지 (Solanum melongena), 오이 (Cucumis sativus), 호박 (Cucurbita moschata), 수박 (Citrullus lanatus), 방울토마토 (Solanum lycopersicum var. cerasiforme), 고구마 (Ipomoea batatas), 들깨 (Perilla frutescens) 종자를 수집하여 일정 기간동안 발아 및 생육시킨 후, 어린잎을 채취하여 동일한 방식으로 DNA 추출 및 PCR을 진행하였을 때 (Fig. 7), 대마 이외의 타 식물체에서는 100 bp 미만의 primer dimer band가 진하게 발현되었고, 목적 band가 아닌 무작위 밴드가 검출되었으며, 대마에서만 특이적으로 약 200 bp의 IS3 목적 band가 검출됨을 확인하였다. SCAR3 목적 band 또한 대마 웅성 개체에서만 검출되어, 이를 통해 분쇄 등의 성상이 변경된 잎에서 타 식물체 중 대마만을 1차 선별하고, 대마의 성별도 동시에 판별할 수 있음을 확인하였다 (Fig. 7B).
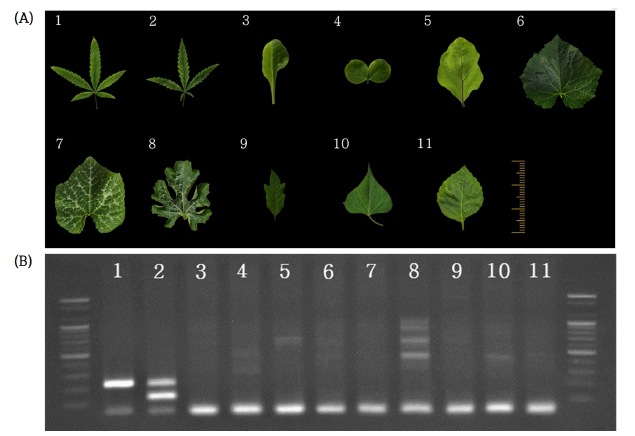
Identification of cannabis among other plants.(A); Plant leaf samples used in the experiment (the 6 ㎝ ruler determine the size of the samples). (B); Various plants using Multiplex-PCR primer set. lane 1; female Cannabis sativa; lane 2; male Cannabis sativa, lane 3; Beta vulgaris var. cicla;, lane 4; Pisum sativum, lane 5; Solanum melongena, lane 6; Cucumis sativus, lane 7; Cucurbita moschata, lane 8; Citrullus lanatus, lane 9; Solanum lycopersicum var. cerasiforme, lane 10; Ipomoea batatas, and lane 11; Perilla frutescens.
향후 SCAR3 프라이머셋과 IS3 프라이머셋을 이용한 multiplex-PCR 마커는 영양생장기의 유식물체 뿐만 아니라 상업적으로 생산된 자성 종자의 genomic DNA을 대상으로 한 성판별에도 활용할 수 있을 것이며, 직접 재배하지 않아도 pollen의 혼입에 따른 종자의 성별을 판별하여 고수율의 칸나비노이드 생산을 위한 원료 식물의 자성화도 평가를 용이하게함으로써 의약품이 원료가 되는 대마식물체의 자성종자 quality control에도 적용 가능할 것으로 생각된다. 또한, PCR의 정상 반응 여부까지도 동시에 판별할 수 있어 매우 높은 유용성을 나타낸다고 하겠다.
Acknowledgments
본 연구는 과학기술정보통신부(과제번호: 2021-DD-UP-0379) 연구과제 및 한국연구재단 4단계 BK21 프로그램의 지원에 의해 이루어진 것으로 이에 감사드립니다.
승인 정보
본 연구에서 사용된 식물체는 서울지방식품의약품안전청의 마약류 학술연구자 승인(허가번호: 서울-제1806)과 마약류 원료물질취급(일반행위금지의예외) 승인(마약정책과-4789호)을 바탕으로 수집, 재배 및 사용, 분석되었습니다.
References
-
Alstrom-Rapaport C, Lascoux M, Wang YC, Roberts G and Tuskan GA. (1998). Identification of a RAPD marker linked to sex determination in the basket willow(Salix viminalis L.). Journal of Heredity. 89:44-49.
[https://doi.org/10.1093/jhered/89.1.44]

-
Aryal R and Ming R. (2014). Sex determination in flowering plants: Papaya as a model system. Plant Science. 217:56-62.
[https://doi.org/10.1016/j.plantsci.2013.10.018]

-
Atalay S, Jarocka-Karpowicz I and Skrzydlewska E. (2019). Antioxidative and anti-inflammatory properties of cannabidiol. Antioxidants. 9:21. https://www.mdpi.com/2076-3921/9/1/21, (cited by 2023 March 21).
[https://doi.org/10.3390/antiox9010021]

-
Bai Q, Ma Z, Zhang Y, Su S and Leng P. (2019). The sex expression and sex determining mechanism in Pistacia species. Breeding Science, 69:205-214.
[https://doi.org/10.1270/jsbbs.18167]

-
Boggs DL, Nguyen JD, Morgenson D, Taffe MA and Ranganathan M. (2018). Clinical and preclinical evidence for functional interactions of cannabidiol and Δ9-tetrahydrocannabinol. Neuropsychopharmacology. 43: 142-154.
[https://doi.org/10.1038/npp.2017.209]

-
Chesney E, Oliver D and McGuire P. (2022). Cannabidiol(CBD) as a novel treatment in the early phases of psychosis. Psychopharmacology. 1179-1190.
[https://doi.org/10.1007/s00213-021-05905-9]

- Consroe P and Wolkin A. (1977). Cannabidiol--antiepileptic drug comparisons and interactions in experimentally induced seizures in rats. Journal of Pharmacology and Experimental Therapeutics. 201:26-32.
-
Crippa JA, Guimarães FS, Campos AC and Zuardi AW. (2018). Translational investigation of the therapeutic potential of cannabidiol (CBD): Toward a new age. Frontiers in Immunology. 9:2009. https://www.frontiersin.org/articles/10.3389/fimmu.2018.02009/full, (cited by 2023 March 21).
[https://doi.org/10.3389/fimmu.2018.02009]

-
de Backer B, Maebe K, Verstraete AG and Charlier C. (2012). Evolution of the content of THC and other major cannabinoids in drug-type cannabis cuttings and seedlings during growth of plants. Journal of Forensic Sciences. 57:918-922.
[https://doi.org/10.1111/j.1556-4029.2012.02068.x]

-
Doh EJ, Lee G, Yun YJ, Kang LW, Kim ES, Lee MY and Oh SE. (2019). DNA markers to discriminate Cannabis sativa L. 'Cheungsam' with low tetrahydrocannabinol(THC) content from other South Korea cultivars based on the nucleotide sequences of tetrahydrocannabinolic acid synthase and putative 3-Ketoacyl-CoA synthase genes. Evidence-Based Complementary and Alternative Medicine. 2019:8121796. https://www.hindawi.com/journals/ecam/2019/8121796/, (cited by 2023 March 21).
[https://doi.org/10.1155/2019/8121796]

-
D'Souza DC, Abi-Saab WM, Madonick S, Forselius-Bielen K, Doersch A, Braley G, Gueorguieva R, Cooper TB and Krystal JH. (2005). Delta-9-tetrahydrocannabinol effects in schizophrenia: Implications for cognition, psychosis, and addiction. Biological Psychiatry. 57:594-608.
[https://doi.org/10.1016/j.biopsych.2004.12.006]

-
ElSohly MA, Radwan MM, Gul W, Chandra S and Galal A. (2017). Phytochemistry of Cannabis sativa L. Phytocannabinoids. Springer. Berlin, Germany. p.1-36.
[https://doi.org/10.1007/978-3-319-45541-9_1]

-
Flajšman M, Slapnik M and Murovec J. (2021). Production of Feminized Seeds of High CBD Cannabis sativa L. by Manipulation of Sex Expression and Its Application to Breeding. Frontiers in Plant Science. 12:718092. https://www.frontiersin.org/articles/10.3389/fpls.2021.718092/full, (cited by 2023 March 21).
[https://doi.org/10.3389/fpls.2021.718092]

-
Freeman D, Dunn G, Murray RM, Evans N, Lister R, Antley A, Slater M, Godlewska B, Cornish R, Williams J, Simplicio MD, Igoumenou A, Brenneisen R, Tunbridge EM, Harrison PJ, Harmer CJ, Cowen P and Morrison PD. (2015). How cannabis causes paranoia: Using the intravenous administration of Δ9-tetrahydrocannabinol(THC) to identify key cognitive mechanisms leading to paranoia. Schizophrenia Bulletin. 41:391-399.
[https://doi.org/10.1093/schbul/sbu098]

-
Go YY, Kim SR, Kim DY, Chae SW and Song JJ. (2020). Cannabidiol enhances cytotoxicity of anti-cancer drugs in human head and neck squamous cell carcinoma. Scientific Reports. 10:20622. https://www.nature.com/articles/s41598-020-77674-y, (cited by 2023 March 21).
[https://doi.org/10.1038/s41598-020-77674-y]

-
Guimarães FS, Chiaretti TM, Graeff FG and Zuardi AW. (1990). Antianxiety effect of cannabidiol in the elevated plus-maze. Psychopharmacology. 100:558-559.
[https://doi.org/10.1007/BF02244012]

-
Gupta SK, Charpe A, Koul S, Haque QMR and Prabhu KV. (2006). Development and validation of SCAR markers co-segregating with an Agropyron elongatum derived leaf rust resistance gene Lr24 in wheat. Euphytica. 150:233-240.
[https://doi.org/10.1007/s10681-006-9113-8]

-
Heikrujam M, Sharma K, Prasad M and Agrawal V. (2015). Review on different mechanisms of sex determination and sex-linked molecular markers in dioecious crops: A current update. Euphytica. 201:161-194.
[https://doi.org/10.1007/s10681-014-1293-z]

-
Hindley G, Beck K, Borgan F, Ginestet CE, McCutcheon R, Kleinloog D, Ganesh S, Radhakrishnan R, D'Souza DC and Howes OD. (2020). Psychiatric symptoms caused by cannabis constituents: A systematic review and meta-analysis. The Lancet Psychiatry. 7:344-353.
[https://doi.org/10.1016/S2215-0366(20)30074-2]

-
Huh MK. (2015). An overview for molecular markers in plants. Journal of Life Science. 25:839-848.
[https://doi.org/10.5352/JLS.2015.25.7.839]

-
Jo IH, Kim YC, Kim DH, Kim KH, Hyun TK, Ryu H and Bang KH. (2017). Applications of molecular markers in the discrimination of Panax species and Korean ginseng cultivars (Panax ginseng). Journal of Ginseng Research. 41:444-449.
[https://doi.org/10.1016/j.jgr.2016.09.001]

-
Khampila J, Lertrat K, Saksirirat W, Sanitchon J, Muangsan N and Theerakulpisut P. (2008). Identification of RAPD and SCAR markers linked to northern leaf blight resistance in waxy corn(Zea mays var. ceratina). Euphytica. 164:615-625.
[https://doi.org/10.1007/s10681-008-9647-z]

-
Kim MK, Kim JH, Lee MS, Jo NS, Park SI, Gil JS, Yeruult E, Oh HK, Lee KH, Kim HB, Lee MS and Lee Y. (2021). Development of insertion or deletion markers to distinguish Korean jujube cultivas. Korean Journal of Medicinal Crop Science. 29:282-292.
[https://doi.org/10.7783/KJMCS.2021.29.4.282]

-
Kim WJ, Noh S, Choi G and Moon BC. (2022). Rapid identification of Paeoniae radix and moutan radicis cortex using a SCAR marker-based conventional PCR assay. Plants. 11:2870. https://www.mdpi.com/2223-7747/11/21/2870, (cited by 2023 March 21).
[https://doi.org/10.3390/plants11212870]

-
Koltai H and Shalev N. (2022). Anti-cancer activity of Cannabis sativa phytocannabinoids: Molecular mechanisms and potential in the fight against ovarian cancer and stem cells. Cancers. 14:4299. https://www.mdpi.com/2072-6694/14/17/4299, (cited by 2023 March 21).
[https://doi.org/10.3390/cancers14174299]

- Lacombe JP. (1980). Discrimination of sexes as a function of early vegetative processes in dioecious hemp(Cannabis sativa L.). Physiologie Vegetale. 18:419-430.
- Lim JH, Jin DC, Sung JS, Bang KH, Kim OT, Cha SW and Park HW. (2007). Discrimination of Astragalus membranaceus(Fisch) Bunge from A. membranaceus(Fisch) Bunge var. mongholicus(Bunge) with SCAR marker. Korean Journal of Medicinal Crop Science. 15:51-55.
- Linacre A and Thorpe J. (1998). Detection and identification of cannabis by DNA. Forensic Science International. 91:71-76.
- Lubell JD and Brand MH. (2018). Foliar sprays of silver thiosulfate produce male flowers on female hemp plants. HortTechnology. 28:743-747.
- Maliepaard C, Alston FH, van Arkel G, Brown LM, Chevreau E, Dunemann F, Evans KM, Gardiner S, Guilford P, van Heusden AW, Janse J, Laurens F, Lynn JR, Manganaris AG, den Nijis APM, Periam N, Rikkerink E, Roche P, Ryder C, Sansavini S, Schmidt H, Tartarini S, Verhaegh JJ, Vrielink-van Ginkel M and King GJ (1998). Aligning male and female linkage maps of apple(Malus pumila Mill.) using multi-allelic markers. Theoretical and Applied Genetics. 97:60-73.
- Mansouri H, Salari F and Asrar Z. (2013). Ethephon application stimulats cannabinoids and plastidic terpenoids production in Cannabis sativa at flowering stage. Industrial Crops and Products. 46:269-273.
- Mohan Ram HY and Sett R. (1982a). Induction of fertile male flowers in genetically female Cannabis sativa plants by silver nitrate and silver thiosulphate anionic complex. Theoretical and Applied Genetics. 62:369-375.
- Mohan Ram HY and Sett R. (1982b). Modification of growth and sex expression in Cannabis sativa by aminoethoxyvinylglycine and ethephon. Zeitschrift Für Pflanzenphysiologie. 105:165-172.
- Moliterni VMC, Cattivelli L, Ranalli P and Mandolino G. (2004). The sexual differentiation of Cannabis sativa L.: A morphological and molecular study. Euphytica. 140:95-106.
- Naqvi NI and Chattoo BB. (1996). Development of a sequence characterized amplified region(SCAR) based indirect selection method for a dominant blast-resistance gene in rice. Genome. 39:26-30.
- Ohmori T, Murata M and Motoyoshi F. (1996). Molecular characterization of RAPD and SCAR markers linked to the Tm-1 locus in tomato. Theoretical and Applied Genetics. 92:151-156.
- Paran I and Michelmore RW. (1993). Development of reliable PCR-based markers linked to downy mildew resistance genes in lettuce. Theoretical and Applied Genetics. 85:985-993.
- Park CG, Bang KH, Kim OT, Jin DC, Kim DH, Sung JS, Seong NS, Park HW and Lee SC. (2007). Development of SCAR marker for discriminating between violet flowered lines and white flowered lines in Chinese bellflower. Korean Journal of Medicinal Crop Science. 15:1-5.
- Rodriguez-Morrison V, Llewellyn D and Zheng Y. (2021). Cannabis yield, potency, and leaf photosynthesis respond differently to increasing light levels in an indoor environment. Frontiers in Plant Science. 12:646020. https://www.frontiersin.org/articles/10.3389/fpls.2021.646020/full, (cited by 2023 March 21).
- Rodziewicz P, Loroch S, Marczak Ł, Sickmann A and Kayser O. (2019). Cannabinoid synthases and osmoprotective metabolites accumulate in the exudates of Cannabis sativa L. glandular trichomes. Plant Science. 284:108-116.
- Russo EB, Jiang HE, Li X, Sutton A, Carboni A, del Bianco F, Mandolino G, Potter DJ, Zhao YX, Bera S, Zhang YB, Lü EG, Ferguson DK, Hueber F, Zhao LC, Liu CJ, Wang YF and Li CS. (2008). Phytochemical and genetic analyses of ancient cannabis from Central Asia. Journal of Experimental Botany. 59:4171-4182.
- Ryu BR, Islam MJ, Azad MOK, Go EJ, Rahman MH, Rana MS, Lim YS and Lim JD. (2021). Conversion characteristics of some major cannabinoids from hemp(Cannabis sativa L.) raw materials by new rapid simultaneous analysis method. Molecules. 26:4113. https://www.mdpi.com/1420-3049/26/14/4113, (cited by 2023 March 21).
- Ryu JH, Lyu JI and Bae CH. (2017). Genetic variation and phylogenetic relationship of taraxacum based on chloroplast DNA(trnL-trnF and rps16-trnK) sequences. Korean Journal of Plant Resources. 30:522-534.
- Sakamoto K, Abe T, Matsuyama T, Yoshida S, Ohmido N, Fukui K and Satoh S. (2005). RAPD markers encoding retrotransposable elements are linked to the male sex in Cannabis sativa L. Genome. 48:931-936.
- Salentijn EMJ, Zhang Q, Amaducci S, Yang M and Trindade LM. (2015). New developments in fiber hemp(Cannabis sativa L.) breeding. Industrial Crops and Products. 68:32-41.
- Schaffner JH. (1921). Influence of environment on sexual expression in hemp. Botanical Gazette. 71:197-219.
- Schultes RE, Klein WM, Plowman T and Lockwood TE. (1975). Cannabis: An example of taxonomic neglect. Cannabis and Culture. De Gruyter Academic Publishing. Berlin, Germany. p.21-38.
- Small E and Beckstead HD. (1973). Cannabinoid phenotypes in Cannabis sativa. Nature. 245:147-148.
- Tahir MN, Shahbazi F, Rondeau-Gagné S and Trant JF. (2021). The biosynthesis of the cannabinoids. Journal of Cannabis Research. 3:7. https://jcannabisresearch.biomedcentral.com/articles/10.1186/s42238-021-00062-4, (cited by 2023 March 21).
- Tajik T, Baghaei K, Moghadam VE, Farrokhi N and Salami SA. (2022). Extracellular vesicles of cannabis with high CBD content induce anticancer signaling in human hepatocellular carcinoma. Biomedicine and Pharmacotherapy. 152:113209. https://www.sciencedirect.com/science/article/pii/S0753332222005984, (cited by 2023 March 21).
- Törjék O, Bucherna N, Kiss E, Homoki H, Finta-Korpelová Z, Bócsa I, Nagy I and Heszky LE. (2002). Novel male-specific molecular markers(MADC5, MADC6) in hemp. Euphytica. 127:209-218.
- Toth JA, Stack GM, Cala AR, Carlson CH, Wilk RL, Crawford JL, Viands DR, Philippe G, Smart CD, Rose JKC and Smart LB. (2020). Development and validation of genetic markers for sex and cannabinoid chemotype in Cannabis sativa L. GCB-Bioenergy. 12:213-222.
- Urits I, Gress K, Charipova K, Habib K, Lee D, Lee C, Jung, JW, Kassem H, Cornett E, Paladini A Varrassi G, Kaye AD and Viswanath O. (2020). Use of cannabidiol(CBD) for the treatment of chronic pain. Best Practice and Research Clinical Anaesthesiology, 34:463-477.
- Xu J, Miao H, Wu H, Huang W, Tang R, Qiu M, Wen J, Zhu S and Li Y. (2006). Screening genetically modified organisms using multiplex-PCR coupled with oligonucleotide microarray. Biosensors and Bioelectronics. 22:71-77.
- Zhang J, Yan J, Huang S, Pan G, Chang L, Li J, Zhang C, Tang H, Chen A and Peng D, Biswas A, Zhang C, Zhao L and Li D. (2020). Genetic diversity and population structure of cannabis based on the genome-wide development of simple sequence repeat markers. Frontiers in Genetics. 11:958. https://www.frontiersin.org/articles/10.3389/fgene.2020.00958/full, (cited by 2023 March 21).

